一、代码作用
GSEA (Gene Set Enrichment Analysis,基因集富集分析)是一种重要的生物信息学分析方法,主要用于分析基因表达数据中的基因集变化,评估预定义基因集在特定实验条件下的表现。它能够发现显著改变的生物学通路,识别功能相关的基因组,并揭示潜在的分子机制。GSEA的优势在于考虑整个基因表达谱,无需设置人为阈值,能够发现微小但一致的变化,使结果更具生物学意义。这种分析方法广泛应用于疾病机制研究、药物作用机制探索、生物标志物发现和治疗靶点筛选等领域。分析过程包括准备表达数据、基因排序、计算富集分数、评估统计显著性和结果可视化,最终通过富集分数(ES)、标准化富集分数(NES)、P值和FDR等参数来解读结果。GSEA已成为理解复杂生物学过程的强大工具,特别适合大规模转录组数据的分析研究。
二、具体的代码
remove(list = ls()) ##清空当前环境
# 设置工作目录(请根据需要修改路径)
setwd("F:/生信代码复现/GSEA基因集富集分析") # 替换为您想保存结果的目录路径
# 检查并安装必要的包
if (!requireNamespace("BiocManager", quietly = TRUE)) {
install.packages("BiocManager")
}
if (!requireNamespace("GseaVis", quietly = TRUE)) {
BiocManager::install("GseaVis")
}
# 加载包
library(GseaVis)
# 数据加载
tryCatch({
test_data <- system.file("extdata", "gseaRes.RDS", package = "GseaVis")
gseaRes <- readRDS(test_data)
}, error = function(e) {
stop("数据加载失败: ", e$message)
})
# 定义目标基因
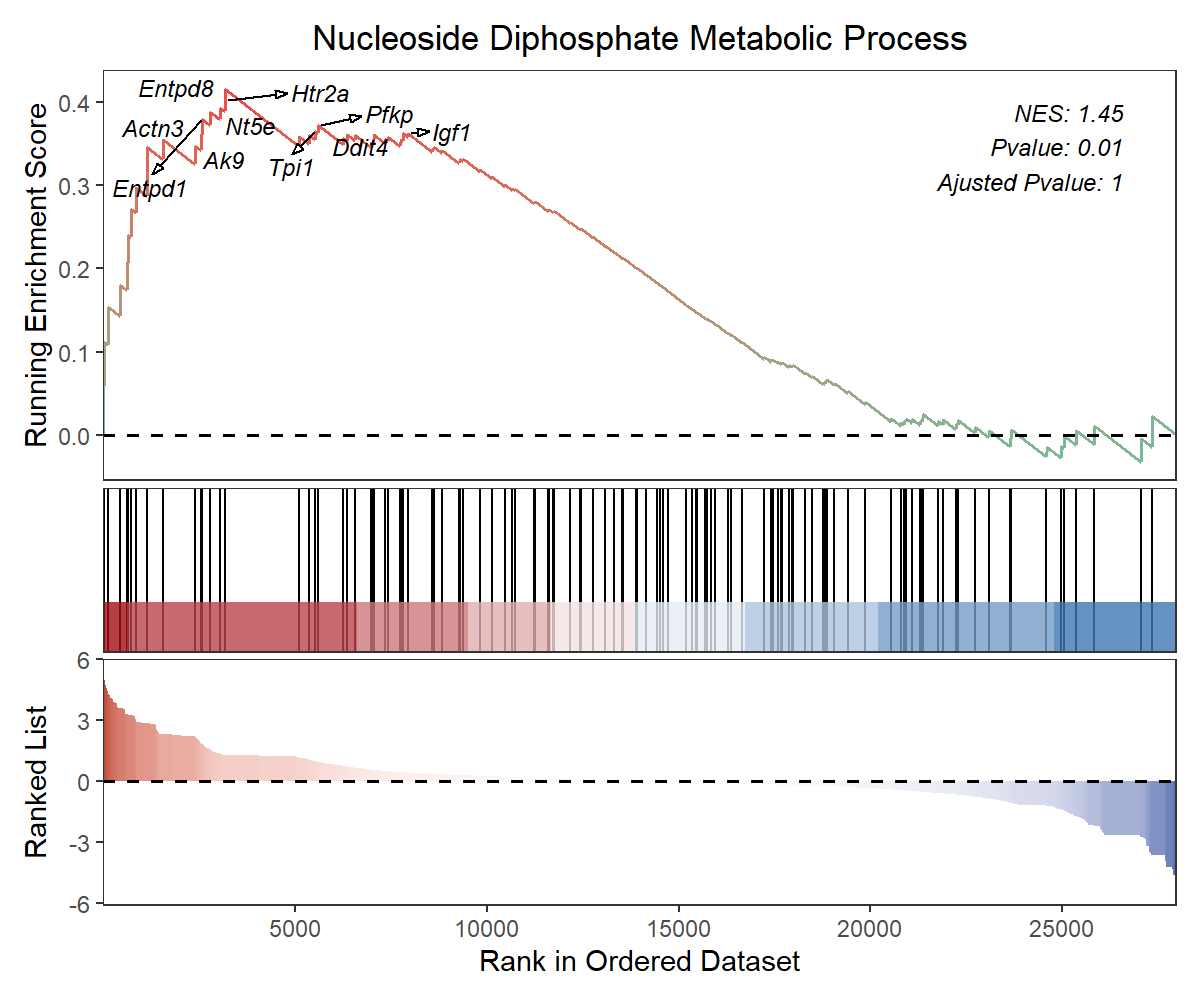
mygene <- c("Entpd8","Htr2a","Nt5e","Actn3","Entpd1",
"Pfkp", "Tpi1","Igf1","Ddit4","Ak9")
# 定义绘图函数,避免代码重复
create_gsea_plot <- function() {
gseaNb(object = gseaRes,
subPlot = 3,
addGene = mygene,
addPval = TRUE,
pvalX = 0.95,
pvalY = 0.8,
pCol = 'black',
pHjust = 1,
geneSetID = 'GOBP_NUCLEOSIDE_DIPHOSPHATE_METABOLIC_PROCESS',
rmSegment = TRUE,
addPoint = TRUE
)
}
# 保存为PDF格式(矢量图)
pdf("GSEA_plot.pdf", width = 10, height = 8)
create_gsea_plot()
dev.off()
# 保存为PNG格式(位图)
png("GSEA_plot.png", width = 1200, height = 1000, res = 150)
create_gsea_plot()
dev.off()
# 保存为TIFF格式(位图,期刊常用)
tiff("GSEA_plot.tiff", width = 1200, height = 1000, res = 150)
create_gsea_plot()
dev.off()
三、复现出来的图片